jorgelb24-web
Actividad dirigida 4
Esta es la actividad dirigia 4, en la que mostramos las diversas tablas o gráficos, en este caso con las cifras del Covid-19 tras un análisis de un sitio Web que mantiene actualizado los datos desde el inicio de la pandemia. Esta actividad la realizamos con el Pandas.
Me conecto a la API : https://api.covid19api.com/
Instalación de Pandas
Para inciar el análisis de los datos instalamos el Pandas y posteriormente el numpy para trabajar con facilidad.
!pip install pandas
Defaulting to user installation because normal site-packages is not writeable
Requirement already satisfied: pandas in c:\programdata\anaconda3\lib\site-packages (1.4.2)
Requirement already satisfied: python-dateutil>=2.8.1 in c:\programdata\anaconda3\lib\site-packages (from pandas) (2.8.2)
Requirement already satisfied: numpy>=1.18.5 in c:\programdata\anaconda3\lib\site-packages (from pandas) (1.21.5)
Requirement already satisfied: pytz>=2020.1 in c:\programdata\anaconda3\lib\site-packages (from pandas) (2021.3)
Requirement already satisfied: six>=1.5 in c:\programdata\anaconda3\lib\site-packages (from python-dateutil>=2.8.1->pandas) (1.16.0)
pip install numpy
Defaulting to user installation because normal site-packages is not writeable
Requirement already satisfied: numpy in c:\programdata\anaconda3\lib\site-packages (1.21.5)
Note: you may need to restart the kernel to use updated packages.
Importar Pandas
Para trabajar con más facilidad, importamos Pandas a pd, para que la nomeclatura al momento de establece run código sea más práctica.
import pandas as pd
Selección de las ciudades con Covid19
Luego que le dimos importar Pandas, procedemos a seleccionar desde el sitio Web de API, las ciudades con Covid19.
url = 'https://api.covid19api.com/countries'
url
'https://api.covid19api.com/countries'
Data Frame
Para poder reflejar los datos de las ciudades, creamos un data frame, en el que a través de Pandas llamaremos para su visualización. y el comando es: df = pd.read_json(url)
df = pd.read_json(url)
df
| Country | Slug | ISO2 | |
|---|---|---|---|
| 0 | Gibraltar | gibraltar | GI |
| 1 | Oman | oman | OM |
| 2 | France | france | FR |
| 3 | Jersey | jersey | JE |
| 4 | Mali | mali | ML |
| ... | ... | ... | ... |
| 243 | Puerto Rico | puerto-rico | PR |
| 244 | Papua New Guinea | papua-new-guinea | PG |
| 245 | Saint Pierre and Miquelon | saint-pierre-and-miquelon | PM |
| 246 | Timor-Leste | timor-leste | TL |
| 247 | Montenegro | montenegro | ME |
248 rows × 3 columns
Visualización de datos
A continuación procedo a mostrar la vizualización de los datos, en la que mostraremos resutaldos de países como España y Panamá, para psoteriormente ver sus gráficos y ver el el desarrollo del virus. De igual manera haremos comparación de estos dos países. Adicional a ello hemos creado una visualización de los países Centroamericanos y su respectiva gráfica comparativa
España
Los datos de Esapaña son mostrados a continuación: Primero hacemos el data frame de este país, para conocer la posición y posteriormente mediante la url procedemos hacer el llamado de los datos desde el primer día de la pandemia hasta la fecha actual. esto lo haremos con: df[df[‘Country’] == ‘Spain’]
df[df['Country'] == 'Spain']
| Country | Slug | ISO2 | |
|---|---|---|---|
| 141 | Spain | spain | ES |
url_rt_es = 'https://api.covid19api.com/country/spain/status/confirmed/live'
df_rt_es = pd.read_json(url_rt_es)
df_rt_es
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 892 | Spain | ES | 40.46 | -3.75 | 12818184 | confirmed | 2022-07-02 00:00:00+00:00 | |||
| 893 | Spain | ES | 40.46 | -3.75 | 12818184 | confirmed | 2022-07-03 00:00:00+00:00 | |||
| 894 | Spain | ES | 40.46 | -3.75 | 12818184 | confirmed | 2022-07-04 00:00:00+00:00 | |||
| 895 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-06 00:00:00+00:00 |
897 rows × 10 columns
Con este data frame mostramos la cabecera de los resultados en España
df_rt_es.head()
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-26 00:00:00+00:00 |
Con este otro data frame se muestra la parte final de la tabla.
df_rt_es.tail()
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 892 | Spain | ES | 40.46 | -3.75 | 12818184 | confirmed | 2022-07-02 00:00:00+00:00 | |||
| 893 | Spain | ES | 40.46 | -3.75 | 12818184 | confirmed | 2022-07-03 00:00:00+00:00 | |||
| 894 | Spain | ES | 40.46 | -3.75 | 12818184 | confirmed | 2022-07-04 00:00:00+00:00 | |||
| 895 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-06 00:00:00+00:00 |
Gráfica
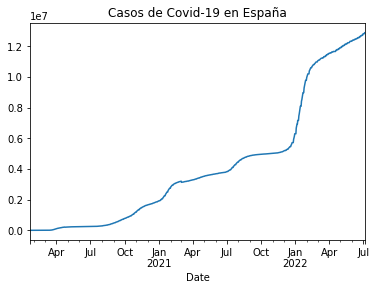
Para mostrar los datos y observar el comportamiento, hemos creado una gráfica y dstacaremos a través de otro data frame la fecha y los casos. En este punto mediante el comando casos_es.plot() plotearemos los datos para que se visualicen los resultados. A esto le colcoamos el título de la gráfica para su identificación.
casos_es = df_rt_es.set_index('Date')['Cases']
casos_es.plot(title="Casos de Covid-19 en España")
<AxesSubplot:title={'center':'Casos de Covid-19 en España'}, xlabel='Date'>
Panamá
En el caso de Panamá, procedimos a realizar el mismo paso que en España.
- Creamos un data frame de la ciudad para concoer su posición
- Hicimos la sellección de los datos a través del url de API.
- Creamos otro data frame para hacer el llaamdao a través de Pandas de la información del url
- Procedimos a visualizar el gráfico para conocer el comprotamiento del virus desde el día 1 a la actualidad. igual manera lo ploteamos para que se posteara.
Paso 1
df[df['Country'] == 'Panama']
| Country | Slug | ISO2 | |
|---|---|---|---|
| 190 | Panama | panama | PA |
Paso 2
url_casos_pa = 'https://api.covid19api.com/country/panama/status/confirmed/live'
Paso 3
df_rt_pa = pd.read_json(url_casos_pa)
df_rt_pa
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 893 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-03 00:00:00+00:00 | |||
| 894 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-04 00:00:00+00:00 | |||
| 895 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-07 00:00:00+00:00 |
898 rows × 10 columns
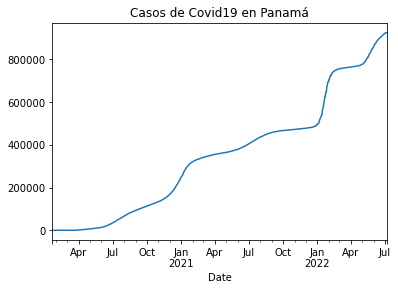
Paso 4
casos_pa = df_rt_pa.set_index('Date')['Cases']
casos_pa.plot(title="Casos de Covid19 en Panamá")
<AxesSubplot:title={'center':'Casos de Covid19 en Panamá'}, xlabel='Date'>
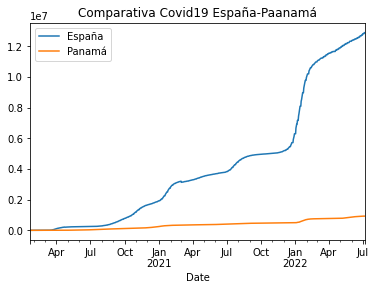
Ploteamos dos países
En esta ocasión hicimos la comparación de los casos de España y panamá, el cual plteamos y mostramos la gráfica comparativa sobre el comprtamiento del virus desde el incio de la pandemia.
Pasos:
- Mediante pa_vs_es = pd.concat([casos_es,casos_pa],axis=1) hacemos el llamado de la selección de la selección de ambas tablas.
- Le colocamos el nombre para identificarla.
pa_vs_es = pd.concat([casos_es,casos_pa],axis=1)
pa_vs_es
| Cases | Cases | |
|---|---|---|
| Date | ||
| 2020-01-22 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-23 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-24 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-25 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-26 00:00:00+00:00 | 0.0 | 0 |
| ... | ... | ... |
| 2022-07-03 00:00:00+00:00 | 12818184.0 | 925254 |
| 2022-07-04 00:00:00+00:00 | 12818184.0 | 925254 |
| 2022-07-05 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-06 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-07 00:00:00+00:00 | NaN | 925254 |
898 rows × 2 columns
En este paso procedemos a identicar cada columna para saber los resultados de cada país, ya que en la tabla anterior solo nos mostraba la variable casos, sin especificar a qué país correspondía cada columna.
pa_vs_es.columns = ['España', 'Panamá']
pa_vs_es
| España | Panamá | |
|---|---|---|
| Date | ||
| 2020-01-22 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-23 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-24 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-25 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-26 00:00:00+00:00 | 0.0 | 0 |
| ... | ... | ... |
| 2022-07-03 00:00:00+00:00 | 12818184.0 | 925254 |
| 2022-07-04 00:00:00+00:00 | 12818184.0 | 925254 |
| 2022-07-05 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-06 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-07 00:00:00+00:00 | NaN | 925254 |
898 rows × 2 columns
Gráfica de comparación
Mediante el ploteó, procedimos a realizar la gráfica comparativa en la que se muestra el comportamiento del virus en ambos países y lo hicimos con el comando: pa_vs_es.plot()
pa_vs_es.plot(title="Comparativa Covid19 España-Paanamá")
<AxesSubplot:title={'center':'Comparativa Covid19 España-Paanamá'}, xlabel='Date'>
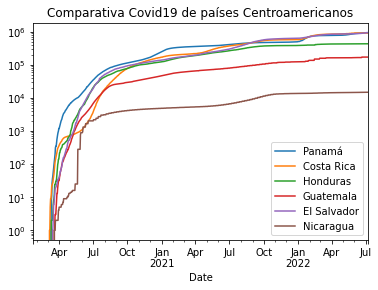
Comparación de países Centroamericanos
En esta ocasión, realizamos una comparación del comprotamiento del virus Covid19, en los paises Centroamericanos y lo hicimos tal cual realizamos con España y Panamá.
- Primero dividí los casos de cada país, en este caso de Costa Rica, El Salvado, Honduras, Nicaragua y Gautemala, para posteriormente realizar la comparación.
- Los casos de cada país lo hice con el url de API.
- Hice el llamado a través de Pandas.
- Hice el plot del gráfico.
Costa Rica
url_casos_cr = 'https://api.covid19api.com/country/costa-rica/status/confirmed/live'
df_rt_cr = pd.read_json(url_casos_cr)
casos_cr = df_rt_cr.set_index('Date')['Cases']
casos_cr.plot(title="Casos de Covid19 en Costa Rica")
<AxesSubplot:title={'center':'Casos de Covid19 en Costa Rica'}, xlabel='Date'>
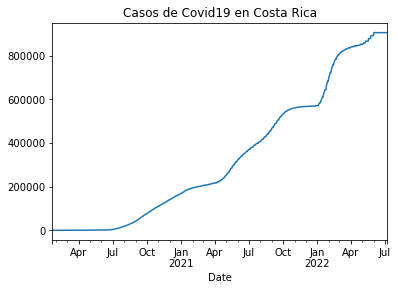
Honduras
url_casos_hnd = 'https://api.covid19api.com/country/honduras/status/confirmed/live'
df_rt_hnd = pd.read_json(url_casos_hnd)
casos_hnd = df_rt_hnd.set_index('Date')['Cases']
casos_hnd.plot(title="Casos de Covid19 en Honduras")
<AxesSubplot:title={'center':'Casos de Covid19 en Honduras'}, xlabel='Date'>
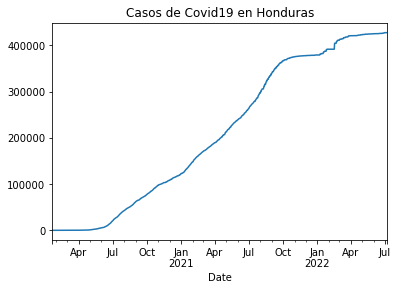
Guatemala
url_casos_guat = 'https://api.covid19api.com/country/guatemala/status/confirmed/live'
df_rt_guat = pd.read_json(url_casos_guat)
casos_guat = df_rt_guat.set_index('Date')['Cases']
casos_guat.plot(title="Casos de Covid19 en Guatemala")
<AxesSubplot:title={'center':'Casos de Covid19 en Guatemala'}, xlabel='Date'>
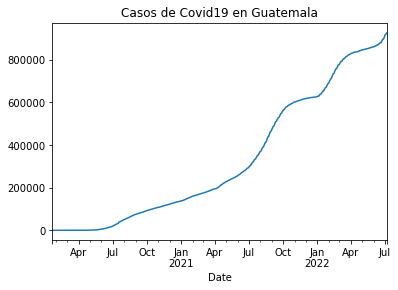
El Salvador
url_casos_elsalv = 'https://api.covid19api.com/country/el-salvador/status/confirmed/live'
df_rt_elsalv = pd.read_json(url_casos_elsalv)
casos_elsalv = df_rt_elsalv.set_index('Date')['Cases']
casos_elsalv.plot(title="Casos de Covid19 en El Salvador")
<AxesSubplot:title={'center':'Casos de Covid19 en El Salvador'}, xlabel='Date'>
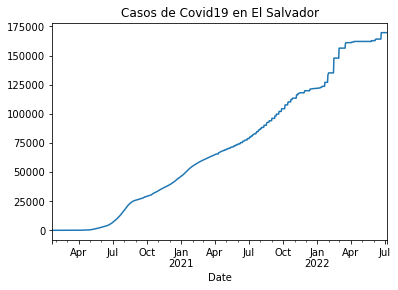
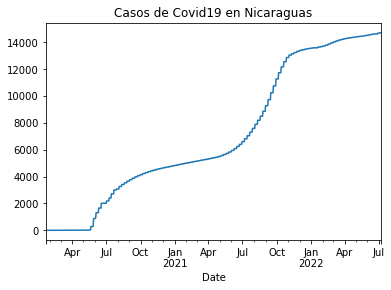
Nicaragua
url_casos_ni = 'https://api.covid19api.com/country/nicaragua/status/confirmed/live'
df_rt_ni = pd.read_json(url_casos_ni)
casos_ni = df_rt_ni.set_index('Date')['Cases']
casos_ni.plot(title="Casos de Covid19 en Nicaraguas")
<AxesSubplot:title={'center':'Casos de Covid19 en Nicaraguas'}, xlabel='Date'>
Comparación de países Centroamericanos
df_ca = pd.concat([casos_pa,casos_cr,casos_hnd,casos_elsalv,casos_guat,casos_ni],axis=1)
df_ca
| Cases | Cases | Cases | Cases | Cases | Cases | |
|---|---|---|---|---|---|---|
| Date | ||||||
| 2020-01-22 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-23 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-24 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-25 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-26 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| ... | ... | ... | ... | ... | ... | ... |
| 2022-07-03 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 920294 | 14690 |
| 2022-07-04 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 921146 | 14690 |
| 2022-07-05 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 922340 | 14690 |
| 2022-07-06 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 927473 | 14721 |
| 2022-07-07 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 927473 | 14721 |
898 rows × 6 columns
df_ca.columns = ['Panamá','Costa Rica','Honduras','Guatemala','El Salvador','Nicaragua']
df_ca
| Panamá | Costa Rica | Honduras | Guatemala | El Salvador | Nicaragua | |
|---|---|---|---|---|---|---|
| Date | ||||||
| 2020-01-22 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-23 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-24 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-25 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-26 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| ... | ... | ... | ... | ... | ... | ... |
| 2022-07-03 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 920294 | 14690 |
| 2022-07-04 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 921146 | 14690 |
| 2022-07-05 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 922340 | 14690 |
| 2022-07-06 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 927473 | 14721 |
| 2022-07-07 00:00:00+00:00 | 925254 | 904934 | 427718 | 169646 | 927473 | 14721 |
898 rows × 6 columns
df_ca.plot(title="Comparativa Covid19 de países Centroamericanos", logy=True)
<AxesSubplot:title={'center':'Comparativa Covid19 de países Centroamericanos'}, xlabel='Date'>