sergiors29-web
Actividad Dirigida 4
La última actividad dirigida del módulo de Periodismo de Datos II consistía en la creación de un cuaderno de Jupyter en la aplicación del mismo nombre. Desde ahí tendríamos que ejecutar códigos con Python y pandas para la presentación y visualización de datos del Covid-19 en España, Panamá y Centroamérica.
En primer lugar, desde el Jupyter Notebook se creó un archivo al que se denominó ad4.ipynb donde nos conectamos a la API https://covid19api.com/
Instalamos la libreria de “pandas” con pip comando, que funciona para la gestión de paquetes utilizado para instalar y administrar paquetes de software escritos en Python.
!pip install pandas
Requirement already satisfied: pandas in c:\users\sergio rivera\anaconda3\lib\site-packages (1.4.2)
Requirement already satisfied: numpy>=1.18.5 in c:\users\sergio rivera\anaconda3\lib\site-packages (from pandas) (1.21.5)
Requirement already satisfied: python-dateutil>=2.8.1 in c:\users\sergio rivera\anaconda3\lib\site-packages (from pandas) (2.8.2)
Requirement already satisfied: pytz>=2020.1 in c:\users\sergio rivera\anaconda3\lib\site-packages (from pandas) (2021.3)
Requirement already satisfied: six>=1.5 in c:\users\sergio rivera\anaconda3\lib\site-packages (from python-dateutil>=2.8.1->pandas) (1.16.0)
Una vez instaladas las librerias de pandas, usamos import para tener acceso al codigo de pandas e importarlo como pd.
import pandas as pd
Se utilizó url para vincular la dirección del sitio web desde donde se obtendrían los datos para la presentación de visualizaciones. Comprobamos que con introducir url la variable está relacionada automáticamente con esta dirección de la API.
url = 'https://api.covid19api.com/countries'
url
'https://api.covid19api.com/countries'
Con df se genera una orden para que pd (pandas) lea la informacion contenida en la url con lenguaje Json. Inmediatamente vemos que al activarlo, se genera una tabla general con todos los datos existentes. Esta parte sirvió como ejemplo para el desarrollo de las otras tareas.
df = pd.read_json(url)
df
| Country | Slug | ISO2 | |
|---|---|---|---|
| 0 | Gibraltar | gibraltar | GI |
| 1 | Oman | oman | OM |
| 2 | France | france | FR |
| 3 | Jersey | jersey | JE |
| 4 | Mali | mali | ML |
| ... | ... | ... | ... |
| 243 | Puerto Rico | puerto-rico | PR |
| 244 | Papua New Guinea | papua-new-guinea | PG |
| 245 | Saint Pierre and Miquelon | saint-pierre-and-miquelon | PM |
| 246 | Timor-Leste | timor-leste | TL |
| 247 | Montenegro | montenegro | ME |
248 rows × 3 columns
Casos Covid-19 en España
Con la variable para dataframe y == le pedimos al sistema que nos genere la posición en la lista para España, con su respectiva nomenclatura, la cual nos ayudaría para la designación de nombres a las variables y funciones a usar en pasos posteriores.
df[df['Country'] == 'Spain']
| Country | Slug | ISO2 | |
|---|---|---|---|
| 141 | Spain | spain | ES |
Para el ejercicio se comenzó utilizando la variable url_rt_es para denominar al sitio web de donde se obtendrian los datos del Covid-19 para España. Con el uso de pd.read_json + la url_rt_es entre parentesis () se imprime una tabla con los datos existentes en la API para Espa;a.
url_rt_es = 'https://api.covid19api.com/country/spain/status/confirmed/live'
df_rt_es = pd.read_json(url_rt_es)
df_rt_es
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 894 | Spain | ES | 40.46 | -3.75 | 12818184 | confirmed | 2022-07-04 00:00:00+00:00 | |||
| 895 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-07 00:00:00+00:00 | |||
| 898 | Spain | ES | 40.46 | -3.75 | 12973615 | confirmed | 2022-07-08 00:00:00+00:00 |
899 rows × 10 columns
Con el uso de .head() se logra imprimer la cabecera de la lista.
df_rt_es.head()
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Spain | ES | 40.46 | -3.75 | 0 | confirmed | 2020-01-26 00:00:00+00:00 |
Con la implementación de .tail() se pueden imprimir los datos alojados al final de la tabla.
df_rt_es.tail()
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 894 | Spain | ES | 40.46 | -3.75 | 12818184 | confirmed | 2022-07-04 00:00:00+00:00 | |||
| 895 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | Spain | ES | 40.46 | -3.75 | 12890002 | confirmed | 2022-07-07 00:00:00+00:00 | |||
| 898 | Spain | ES | 40.46 | -3.75 | 12973615 | confirmed | 2022-07-08 00:00:00+00:00 |
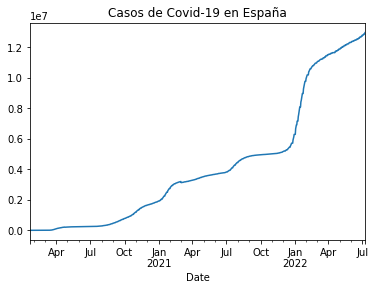
Se le coloca nuevo nombre a las funciones para facilitar su ejecucion. Mas adelante tambien se expone que un index aloja los datos de fechas “Date” mientras que en el otro eje Y se muestra el numero de contagios. Con el uso de .plot() y title se le puede poner titulo a la visualizacion.
casos_es = df_rt_es.set_index('Date')['Cases']
casos_es.plot(title="Casos de Covid-19 en España")
<AxesSubplot:title={'center':'Casos de Covid-19 en España'}, xlabel='Date'>
Casos Covid-19 en Panamá
El mismo procedimiento realizado con los datos de España se vuelven a cumplir para el caso de Panamá. Se identifica la nomenclatura de país para Panamá y se imprime la posición dentro de este listado.
df[df['Country'] == 'Panama']
| Country | Slug | ISO2 | |
|---|---|---|---|
| 190 | Panama | panama | PA |
Con la nueva url se leeran e imprimiran los datos de covid-19 para el pais.
url_rt_pa = 'https://api.covid19api.com/country/panama/status/confirmed/live'
df_rt_pa = pd.read_json(url_rt_pa)
df_rt_pa
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 895 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-07 00:00:00+00:00 | |||
| 898 | Panama | PA | 8.54 | -80.78 | 932710 | confirmed | 2022-07-08 00:00:00+00:00 | |||
| 899 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-09 00:00:00+00:00 |
900 rows × 10 columns
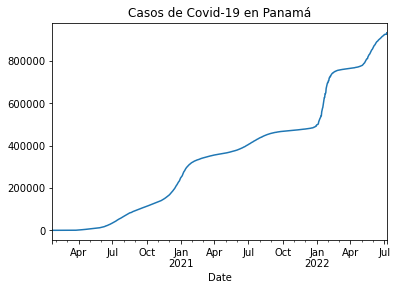
Una vez realizado un vistazo a las cifras de Covid-19 en Panama, usamos las funciones .set_index y .plop() para imprimer los resultados.
casos_pa = df_rt_pa.set_index('Date')['Cases']
casos_pa.plot(title="Casos de Covid-19 en Panamá")
<AxesSubplot:title={'center':'Casos de Covid-19 en Panamá'}, xlabel='Date'>
Ploteamos dos países
De forma previa ya teníamos los datos de Covid-19 en Panamá y España. Como parte de las tareas en clases, se denominaron variables para Panamá casos_pa y España casos_es. Comprobamos que se listaran las cifras para cada país.
casos_pa
Date
2020-01-22 00:00:00+00:00 0
2020-01-23 00:00:00+00:00 0
2020-01-24 00:00:00+00:00 0
2020-01-25 00:00:00+00:00 0
2020-01-26 00:00:00+00:00 0
...
2022-07-05 00:00:00+00:00 925254
2022-07-06 00:00:00+00:00 925254
2022-07-07 00:00:00+00:00 925254
2022-07-08 00:00:00+00:00 932710
2022-07-09 00:00:00+00:00 925254
Name: Cases, Length: 900, dtype: int64
casos_es
Date
2020-01-22 00:00:00+00:00 0
2020-01-23 00:00:00+00:00 0
2020-01-24 00:00:00+00:00 0
2020-01-25 00:00:00+00:00 0
2020-01-26 00:00:00+00:00 0
...
2022-07-04 00:00:00+00:00 12818184
2022-07-05 00:00:00+00:00 12890002
2022-07-06 00:00:00+00:00 12890002
2022-07-07 00:00:00+00:00 12890002
2022-07-08 00:00:00+00:00 12973615
Name: Cases, Length: 899, dtype: int64
Para observar los datos que serían comparados en primera instancia se usó pd.concat con el uso de variables para ambos paises y axis para la muestra de datos en columnas. Previamente se le puso un nombre para facilitar la busqueda de los datos a comparar.
pa_vs_es = pd.concat([casos_es, casos_pa],axis=1)
pa_vs_es
| Cases | Cases | |
|---|---|---|
| Date | ||
| 2020-01-22 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-23 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-24 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-25 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-26 00:00:00+00:00 | 0.0 | 0 |
| ... | ... | ... |
| 2022-07-05 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-06 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-07 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-08 00:00:00+00:00 | 12973615.0 | 932710 |
| 2022-07-09 00:00:00+00:00 | NaN | 925254 |
900 rows × 2 columns
Al colocar .columns se le pone nombre a los datos presentados en la tabla que utilizaremos para realizar el comparativo de casos. Observamos los resultados del uso de estos comandos, en la siguiente presentación.
pa_vs_es.columns = ['España','Panamá']
pa_vs_es
| España | Panamá | |
|---|---|---|
| Date | ||
| 2020-01-22 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-23 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-24 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-25 00:00:00+00:00 | 0.0 | 0 |
| 2020-01-26 00:00:00+00:00 | 0.0 | 0 |
| ... | ... | ... |
| 2022-07-05 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-06 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-07 00:00:00+00:00 | 12890002.0 | 925254 |
| 2022-07-08 00:00:00+00:00 | 12973615.0 | 932710 |
| 2022-07-09 00:00:00+00:00 | NaN | 925254 |
900 rows × 2 columns
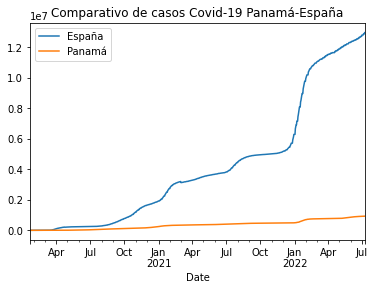
Con el nombre asignado a nuestros datos, sumado a .plot podemos imprimir la gráfica de visualización, donde se nos permite ver el comportamiento de los casos de Covid-19 en España, en comparación con Panamá. En esta parte se le puede colocar un título a la visualización.
pa_vs_es.plot(title="Comparativo de casos Covid-19 Panamá-España")
<AxesSubplot:title={'center':'Comparativo de casos Covid-19 Panamá-España'}, xlabel='Date'>
Comparativo de países de Centroamérica
Se nos pidió realizar un comparativo de las cifras de Covid-19 entre los países de Centroamérica, a saber:
- Guatemala
- El Salvador
- Honduras
- Nicaragua
- Costa Rica
- Panamá
Comenzamos por echar un vistazo a las cifras de Covid-19 para cada país, resaltando que ya conocemos los de Panamá. Para cada uno de los paises de Centroamerica tambien podremos ver una visualizacion, con el uso de un index y un .plot().
url_rt_pa = 'https://api.covid19api.com/country/panama/status/confirmed/live'
df_rt_pa = pd.read_json(url_rt_pa)
df_rt_pa
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Panama | PA | 8.54 | -80.78 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 895 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-07 00:00:00+00:00 | |||
| 898 | Panama | PA | 8.54 | -80.78 | 932710 | confirmed | 2022-07-08 00:00:00+00:00 | |||
| 899 | Panama | PA | 8.54 | -80.78 | 925254 | confirmed | 2022-07-09 00:00:00+00:00 |
900 rows × 10 columns
Guatemala
url_rt_gt = 'https://api.covid19api.com/country/guatemala/status/confirmed/live'
df_rt_gt = pd.read_json(url_rt_gt)
df_rt_gt
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Guatemala | GT | 15.78 | -90.23 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Guatemala | GT | 15.78 | -90.23 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Guatemala | GT | 15.78 | -90.23 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Guatemala | GT | 15.78 | -90.23 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Guatemala | GT | 15.78 | -90.23 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 895 | Guatemala | GT | 15.78 | -90.23 | 922340 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Guatemala | GT | 15.78 | -90.23 | 927473 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | Guatemala | GT | 15.78 | -90.23 | 933259 | confirmed | 2022-07-07 00:00:00+00:00 | |||
| 898 | Guatemala | GT | 15.78 | -90.23 | 939300 | confirmed | 2022-07-08 00:00:00+00:00 | |||
| 899 | Guatemala | GT | 15.78 | -90.23 | 939300 | confirmed | 2022-07-09 00:00:00+00:00 |
900 rows × 10 columns
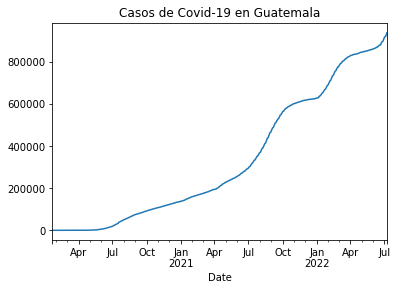
casos_gt = df_rt_gt.set_index('Date')['Cases']
casos_gt.plot(title="Casos de Covid-19 en Guatemala")
<AxesSubplot:title={'center':'Casos de Covid-19 en Guatemala'}, xlabel='Date'>
El Salvador
url_rt_sv = 'https://api.covid19api.com/country/el-salvador/status/confirmed/live'
df_rt_sv = pd.read_json(url_rt_sv)
df_rt_sv
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | El Salvador | SV | 13.79 | -88.9 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | El Salvador | SV | 13.79 | -88.9 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | El Salvador | SV | 13.79 | -88.9 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | El Salvador | SV | 13.79 | -88.9 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | El Salvador | SV | 13.79 | -88.9 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 895 | El Salvador | SV | 13.79 | -88.9 | 169646 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | El Salvador | SV | 13.79 | -88.9 | 169646 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | El Salvador | SV | 13.79 | -88.9 | 169646 | confirmed | 2022-07-07 00:00:00+00:00 | |||
| 898 | El Salvador | SV | 13.79 | -88.9 | 180970 | confirmed | 2022-07-08 00:00:00+00:00 | |||
| 899 | El Salvador | SV | 13.79 | -88.9 | 169646 | confirmed | 2022-07-09 00:00:00+00:00 |
900 rows × 10 columns
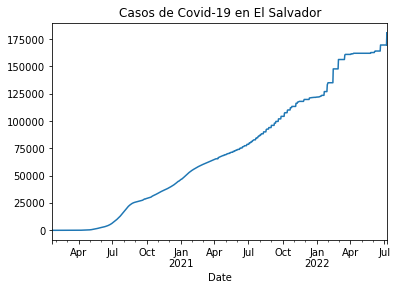
casos_sv = df_rt_sv.set_index('Date')['Cases']
casos_sv.plot(title="Casos de Covid-19 en El Salvador")
<AxesSubplot:title={'center':'Casos de Covid-19 en El Salvador'}, xlabel='Date'>
Honduras
url_rt_hn = 'https://api.covid19api.com/country/honduras/status/confirmed/live'
df_rt_hn = pd.read_json(url_rt_hn)
df_rt_hn
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Honduras | HN | 15.2 | -86.24 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Honduras | HN | 15.2 | -86.24 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Honduras | HN | 15.2 | -86.24 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Honduras | HN | 15.2 | -86.24 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Honduras | HN | 15.2 | -86.24 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 895 | Honduras | HN | 15.2 | -86.24 | 427718 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Honduras | HN | 15.2 | -86.24 | 427718 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | Honduras | HN | 15.2 | -86.24 | 427718 | confirmed | 2022-07-07 00:00:00+00:00 | |||
| 898 | Honduras | HN | 15.2 | -86.24 | 429408 | confirmed | 2022-07-08 00:00:00+00:00 | |||
| 899 | Honduras | HN | 15.2 | -86.24 | 429408 | confirmed | 2022-07-09 00:00:00+00:00 |
900 rows × 10 columns
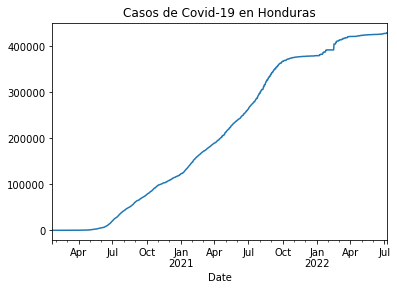
casos_hn = df_rt_hn.set_index('Date')['Cases']
casos_hn.plot(title="Casos de Covid-19 en Honduras")
<AxesSubplot:title={'center':'Casos de Covid-19 en Honduras'}, xlabel='Date'>
Nicaragua
url_rt_ni = 'https://api.covid19api.com/country/nicaragua/status/confirmed/live'
df_rt_ni = pd.read_json(url_rt_ni)
df_rt_ni
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Nicaragua | NI | 12.87 | -85.21 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Nicaragua | NI | 12.87 | -85.21 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Nicaragua | NI | 12.87 | -85.21 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Nicaragua | NI | 12.87 | -85.21 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Nicaragua | NI | 12.87 | -85.21 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 895 | Nicaragua | NI | 12.87 | -85.21 | 14690 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Nicaragua | NI | 12.87 | -85.21 | 14721 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | Nicaragua | NI | 12.87 | -85.21 | 14721 | confirmed | 2022-07-07 00:00:00+00:00 | |||
| 898 | Nicaragua | NI | 12.87 | -85.21 | 14721 | confirmed | 2022-07-08 00:00:00+00:00 | |||
| 899 | Nicaragua | NI | 12.87 | -85.21 | 14721 | confirmed | 2022-07-09 00:00:00+00:00 |
900 rows × 10 columns
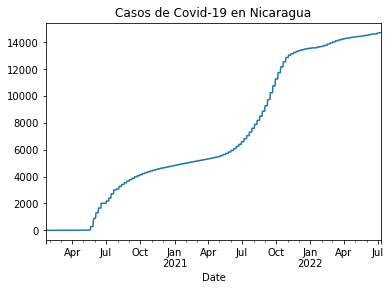
casos_ni = df_rt_ni.set_index('Date')['Cases']
casos_ni.plot(title="Casos de Covid-19 en Nicaragua")
<AxesSubplot:title={'center':'Casos de Covid-19 en Nicaragua'}, xlabel='Date'>
Costa Rica
url_rt_cr = 'https://api.covid19api.com/country/costa-rica/status/confirmed/live'
df_rt_cr = pd.read_json(url_rt_cr)
df_rt_cr
| Country | CountryCode | Province | City | CityCode | Lat | Lon | Cases | Status | Date | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Costa Rica | CR | 9.75 | -83.75 | 0 | confirmed | 2020-01-22 00:00:00+00:00 | |||
| 1 | Costa Rica | CR | 9.75 | -83.75 | 0 | confirmed | 2020-01-23 00:00:00+00:00 | |||
| 2 | Costa Rica | CR | 9.75 | -83.75 | 0 | confirmed | 2020-01-24 00:00:00+00:00 | |||
| 3 | Costa Rica | CR | 9.75 | -83.75 | 0 | confirmed | 2020-01-25 00:00:00+00:00 | |||
| 4 | Costa Rica | CR | 9.75 | -83.75 | 0 | confirmed | 2020-01-26 00:00:00+00:00 | |||
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 895 | Costa Rica | CR | 9.75 | -83.75 | 904934 | confirmed | 2022-07-05 00:00:00+00:00 | |||
| 896 | Costa Rica | CR | 9.75 | -83.75 | 904934 | confirmed | 2022-07-06 00:00:00+00:00 | |||
| 897 | Costa Rica | CR | 9.75 | -83.75 | 904934 | confirmed | 2022-07-07 00:00:00+00:00 | |||
| 898 | Costa Rica | CR | 9.75 | -83.75 | 904934 | confirmed | 2022-07-08 00:00:00+00:00 | |||
| 899 | Costa Rica | CR | 9.75 | -83.75 | 904934 | confirmed | 2022-07-09 00:00:00+00:00 |
900 rows × 10 columns
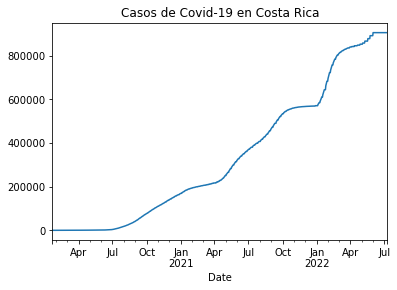
casos_cr = df_rt_cr.set_index('Date')['Cases']
casos_cr.plot(title="Casos de Covid-19 en Costa Rica")
<AxesSubplot:title={'center':'Casos de Covid-19 en Costa Rica'}, xlabel='Date'>
Se listaron las variables para halar los datos de cada país a comparar. En el dataframe hay un index que corresponde a las fechas () “Date”. Lo que se quiere es que se muestren los valores de los casos correspondientes entre [], que corresponde a los “Cases”.
Las visualizaciones se generan con .plot() de acuerdo con la secuencia que se ha tenido con las funciones utilizadas a lo largo del ejercicio.
casos_cr = df_rt_cr.set_index('Date')['Cases']
casos_hn = df_rt_hn.set_index('Date')['Cases']
casos_sv = df_rt_sv.set_index('Date')['Cases']
casos_gt = df_rt_gt.set_index('Date')['Cases']
casos_ni = df_rt_ni.set_index('Date')['Cases']
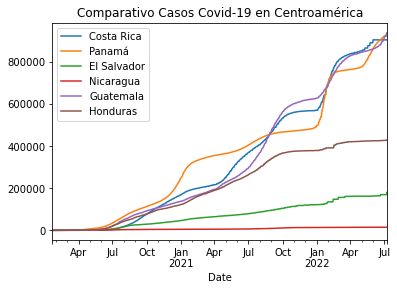
Asignamos el nombre covid_centroamerica a la variable que nos permitirá hacer el comparativo entre los seis países de Centroamérica. Con un pd.concat agrupamos las variables con las que se indentificó a cada país acompañado de axis=1. Con columns se agregaron los nombres para cada columna de datos y se utilizó un .plot() para imprimir la grafica comparativa, donde se pueden observar comportamientos similares en cuanto al movimiento de casos de Covid-19 entre enero de 2020 y julio de 2022.
Con la misma variable covid_centroamerica tenemos acceso a la tabla con los datos organizados para cada uno de los países seleccionados para el análisis y visualización.
covid_centroamerica
| Costa Rica | Panamá | El Salvador | Nicaragua | Guatemala | Honduras | |
|---|---|---|---|---|---|---|
| Date | ||||||
| 2020-01-22 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-23 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-24 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-25 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| 2020-01-26 00:00:00+00:00 | 0 | 0 | 0 | 0 | 0 | 0 |
| ... | ... | ... | ... | ... | ... | ... |
| 2022-07-05 00:00:00+00:00 | 904934 | 925254 | 169646 | 14690 | 922340 | 427718 |
| 2022-07-06 00:00:00+00:00 | 904934 | 925254 | 169646 | 14721 | 927473 | 427718 |
| 2022-07-07 00:00:00+00:00 | 904934 | 925254 | 169646 | 14721 | 933259 | 427718 |
| 2022-07-08 00:00:00+00:00 | 904934 | 932710 | 180970 | 14721 | 939300 | 429408 |
| 2022-07-09 00:00:00+00:00 | 904934 | 925254 | 169646 | 14721 | 939300 | 429408 |
900 rows × 6 columns
covid_centroamerica = pd.concat([casos_cr, casos_pa, casos_sv, casos_ni, casos_gt, casos_hn],axis=1)
covid_centroamerica.columns = ['Costa Rica','Panamá','El Salvador','Nicaragua','Guatemala','Honduras']
covid_centroamerica.plot(title="Comparativo Casos Covid-19 en Centroamérica")
<AxesSubplot:title={'center':'Comparativo Casos Covid-19 en Centroamérica'}, xlabel='Date'>
Una vez culminado el desarrollo de la actividad, se procedió a guardar la misma versión de nuestro archivo inicial y se creó uno con el nombre en terminación .md para luego hacer las respectivas actualizaciones en la terminal de git bash para que se reflejasen en el repositorio remoto.